Mapeando células aos milhares
Professor
da Unicamp participa de pesquisa pioneira na área
da genômica
PAULO
CÉSAR NASCIMENTO
|
|
|
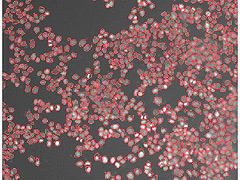
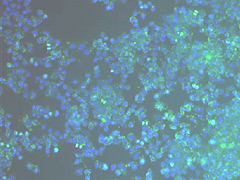
À imagem digital de células captada
e ampliada 200 vezes por microscópio a
partir de amostra (spot) na lâmina de microarray;
em seguida, a mesma imagem depois de processada
pelo software, que delineia e identifica automaticamente
os núcleos das células |
A
experiência de um professor da Unicamp e um
software desenvolvido por docentes da Universidade
e de três outros centros brasileiros de pesquisa
foram decisivos para a conclusão de um estudo
divulgado em outubro pela respeitada Genome Research,
publicação norte-americana voltada à
Genômica. Roberto Lotufo, do Departamento de
Engenharia de Computação e Automação
Industrial da Faculdade de Engenharia Elétrica
e Computação (FEEC) utilizou uma ferramenta
de processamento morfológico de imagens na
análise de células submetidas a um revolucionário
processo conhecido como interferência de RNA
(RNAi), capaz de desligar ou silenciar genes daninhos
e, portanto, potencialmente eficaz no combate a vírus
e tumores (leia texto nesta página).
Conduzida pelo National Human Genome Research Institute,
do National Institutes of Health (NIH), dos Estados
Unidos, a pesquisa da qual Lotufo participou integra
uma série de experimentos pioneiros em genética,
que apontam para a possibilidade de curar câncer
e tratar infecções virais letais usando
a técnica da RNAi.
O trabalho exigia velocidade, qualidade
e precisão na análise de imagens, requisitos
que apenas o software brasileiro foi capaz de cumprir,
conta Lotufo, um dos responsáveis pelo seu
desenvolvimento. O software foi possível graças
à cooperação iniciada na década
de 90 com os professores Gerald Jean Francis Banon,
do Instituto de Pesquisas Espaciais (Inpe), Junior
Barrera, da Universidade de São Paulo (USP)
e mais recentemente com o pesquisador Rubens Campos
Machado, do Centro de Pesquisas Renato Archer (CenPRA).
Durante o desenvolvimento do programa,
inicialmente para apoio ao ensino e pesquisa, o grupo
contou com a colaboração do professor
Edward Dougherty, atual diretor do Laboratório
de Processamento de Sinal Genômico da Texas
A&M University (EUA), uma das autoridades mundiais
em processamento de imagem e análise de dados
genômicos, e consultor do NIH.
“A qualidade do sistema e a
minha experiência prática no seu desenvolvimento
valeram convite do professor Dougherty para que eu
participasse da pesquisa com RNAi junto com ele e
mais dez cientistas”, conta Lotufo, único
brasileiro a assinar o trabalho.
Rigor matemático –
O diferencial do software brasileiro – na verdade,
um conjunto de ferramentas (toolbox) para diferentes
aplicações – está no emprego
de morfologia matemática na análise
de imagem.
Se em Biologia o termo morfologia
refere-se ao estudo da forma e estrutura de plantas
e animais, em processamento digital a expressão
representa o uso de cálculos matemáticos
para a análise da estrutura geométrica
contida em uma imagem.
“Graças a isso, a toolbox
trata problemas de processamento de imagem com um
rigor matemático muito forte e oferece flexibilidade
para um amplo espectro de aplicações,
proporcionando resultados superiores em relação
aos processos que não utilizam morfologia”,
explica o professor da Unicamp, um dos poucos no mundo
que dominam a tecnologia.
Ele também observa que, conforme
a própria denominação, o sistema
não é uma solução pronta
para o usuário final, mas disponibiliza os
recursos para o profissional selecioná-los
e adequá-los para solucionar diferentes problemas
em processamento de imagens, seja para uma aplicação
industrial seja para uma aplicação médica,
como ocorreu no estudo do NIH.
A pesquisa com a RNAi foi realizada
em 2002. Nos três meses em que permaneceu na
Texas A&M University e no instituto norte-americano,
Lotufo responsabilizou-se pelo processamento e análise
de imagens de células depositadas em lâminas
de vidro, geradas em um sofisticado processo conhecido
por microarray, técnica que possibilita a avaliação
simultânea de milhares de genes (leia matéria
à parte).
A dificuldade da empreitada residiu
no fato de que essa tecnologia gera uma quantidade
absurda de dados. No estudo em questão, foram
analisadas 20 mil diferentes imagens de cada lâmina
contendo duas mil amostras (spots) microscópicas
de fragmentos celulares, com aproximadamente três
mil células em cada imagem.
|

|
|
O professor Roberto Lotufo, da FEEC: morfologia
matemática na análise de imagem |
“Conseguimos reduzir o tempo
de processamento de cada imagem para menos de 5 segundos.
Foram a velocidade e o baixo nível de erro
do sistema que permitiram ao grupo colher os resultados
desejados, já que os softwares comerciais inicialmente
procurados não apresentavam nem o desempenho
nem a qualidade de imagem que o estudo exigia”,
conta Lotufo.
Download gratuito –
A tecnologia do processamento morfológico de
imagem está disponível para interessados
em Hands-on Morphological Image Processing, livro
que Lotufo escreveu em co-autoria com Edward Dougherty.
Comercializada por meio da Internet pela Amazon, a
obra oferece a estudantes e profissionais a oportunidade
de experimentar diferentes aplicações
práticas com a coleção de ferramentas
contidas no software.
Uma versão didática
do programa está à disposição
na rede mundial de computadores para download gratuito
no seguinte endereço: http://sourceforge.net/projects/pymorph.
Com esta versão é possível reproduzir
a maioria dos exemplos discutidos no livro, facilitando
a disseminação da técnica apresentada
pelos autores.
****************************************
Técnica
é comparada ao surgimento dos antibióticos
A técnica conhecida como
RNA interferente (RNAi) utiliza moléculas curtas
de RNA (designação do ácido ribonucléico)
para neutralizar genes específicos. De tão
fantástica, a descoberta está sendo
comparada ao desenvolvimento dos antibióticos,
que modificaram radicalmente o modo pelo qual a medicina
tratava infecções bacterianas no século
20.
A estrutura do RNA é semelhante
à do DNA, molécula-código das
características genéticas. Moléculas
de RNA de tamanho normal convertem informações
genéticas em proteínas. Porém,
no início da década de 90, Andrew Fire,
da Universidade Johns Hopkins (EUA), descobriu que
moléculas truncadas de RNA poderiam ser induzidas
para silenciar ou desligar os mesmos genes que tinham
ajudado a gerá-las. Na prática, significava
que genes defeituosos responsáveis por tumores,
ou genes usados por um vírus para se reproduzir,
poderiam ser neutralizados.
Ele nomeou o efeito de “interferência de
RNA” (RNAi) e argumentou que os genes podem ser
silenciados seletivamente, ou seja, a interferência
não resultaria no silenciamento de outros genes
importantes.
A teoria da RNAi como tratamento
médico começou a ser testada no final
da última década. Estudos laboratoriais
conduzidos nos EUA e na Europa demonstraram que células
humanas podem ser modificadas para resistir à
infecção causada pelo vírus da
pólio e do HIV (causador da Aids).
Em um tratamento que utilizasse
a técnica, as células do sangue de um
paciente contaminado seriam retiradas e modificadas
com uso da RNAi para torná-las imunes ao HIV,
o que então tornaria o paciente resistente
à infecção.
No começo deste ano a revista
Science divulgou as primeiras demonstrações
do potencial terapêutico da RNAi contra doenças
em animais. Os imunologistas Judy Lieberman e Premlata
Shankar, da Universidade de Harvard, administraram
uma solução de moléculas curtas
de RNA interferente (siRNA) na cauda de um rato e
protegeu-o contra a hepatite. E em animais que já
estavam doentes, a RNAi reduziu a inflamação
o bastante para permitir a recuperação
do fígado.
Os pesquisadores acreditam que o
processo também poderá ser eficiente
contra câncer e evitar problemas de rejeição
em transplantes.
****************************************
O
que são microarrays
A técnica dos microarrays
permite o estudo confiável da função
e dos padrões de expressão dos genes
tornando-se uma ferramenta poderosa na busca de causadores
genéticos de tumores. Cada spot analisado tem
frações de milímetro e é
fixado na lâmina por um robô, que deposita
cada pontinho de amostra em uma seqüência
e identificação distintas.
Pela comparação simultânea
de milhares de amostras de genes ou fragmentos de
genes de células normais e cancerosas, ou de
células pulmonares sãs e das de um doente,
os cientistas conseguem saber quais genes estão
ativos, quais deixaram de funcionar ou estão
funcionando desordenadamente em cada situação.
Dessa maneira, é possível
identificar em um pulmão com asma, por exemplo,
a presença de uma série de genes que
não são observados no pulmão
normal, e concluir que esses genes devem estar relacionados
com a patologia estudada.
No caso do estudo em que Lotufo participou, os microarrays
são feitos de células vivas depositadas
em cada spot. Foram as imagens destas células,
ampliadas por microscópio, processadas e analisadas
pelo software desenvolvido por Lotufo, que possibilitaram
identificar o número de células que
haviam reagido à presença de moléculas
curtas de RNA interferente (siRNA) no estudo do NIH.
“Existiram células que
responderam positivamente a esse agente e outras não.
Meu trabalho foi encontrar a taxa de expressão
de células que foram silenciadas pelo RNAi
e a das que permaneceram ativas.”







