| |
Software mapeia funções cerebrais
Ferramenta desenvolvida
no IC analisa experimentalmente
imagens de ressonância magnética
ISABEL
GARDENAL
 Graças
à tecnologia do diagnóstico por imagem, o mapeamento das funções
cerebrais pode ser realizado hoje com maior precisão e com
a vantagem de não requerer procedimentos invasivos. Um novo
software denominado Bia (Brain Image Analyzer) – desenvolvido
na Unicamp por Paulo André Vechiatto de Miranda, doutor pelo
Instituto de Computação (IC), e pelo grupo do Laboratório
de Informação Visual (LIV) – está analisando experimentalmente
imagens de ressonância magnética em que se extraem medidas
de estruturas do cérebro, tais como do cerebelo e dos hemisférios,
buscando identificar a sua correlação com determinadas doenças.
Essas imagens são tridimensionais (3D) e contam com um mínimo
de intervenção do usuário, devendo se estender, em breve,
para as estruturas subcorticais (localizadas abaixo do córtex).
“Para que o software chegue à versão final, no entanto, será
preciso estender a atual base de treinamentos, que é de 45
imagens, para cerca de 200 imagens”, relata Miranda. A primeira
versão do software está prevista para fevereiro de 2010. Graças
à tecnologia do diagnóstico por imagem, o mapeamento das funções
cerebrais pode ser realizado hoje com maior precisão e com
a vantagem de não requerer procedimentos invasivos. Um novo
software denominado Bia (Brain Image Analyzer) – desenvolvido
na Unicamp por Paulo André Vechiatto de Miranda, doutor pelo
Instituto de Computação (IC), e pelo grupo do Laboratório
de Informação Visual (LIV) – está analisando experimentalmente
imagens de ressonância magnética em que se extraem medidas
de estruturas do cérebro, tais como do cerebelo e dos hemisférios,
buscando identificar a sua correlação com determinadas doenças.
Essas imagens são tridimensionais (3D) e contam com um mínimo
de intervenção do usuário, devendo se estender, em breve,
para as estruturas subcorticais (localizadas abaixo do córtex).
“Para que o software chegue à versão final, no entanto, será
preciso estender a atual base de treinamentos, que é de 45
imagens, para cerca de 200 imagens”, relata Miranda. A primeira
versão do software está prevista para fevereiro de 2010.
O software resulta da tese
de doutorado de Miranda, recém-defendida no IC, cuja abordagem
foi o reconhecimento e delineamento sinérgicos de objetos
em imagens com aplicações na medicina. A tese foi orientada
pelo professor Alexandre Xavier Falcão. A criação do software
aconteceu no âmbito do projeto temático “Identificação e caracterização
das etiologias, mecanismos de lesão, disfunção neuronal e
defeitos moleculares na epilepsia de lobo temporal mesial
e sua relação com a resposta ao tratamento”, coordenado pela
professora Íscia Lopes Cendes, do Departamento de Genética
da Faculdade de Ciências Médicas (FCM). Também teve a participação
do programa Cinapce (Cooperação Interinstitucional de Apoio
a Pesquisas sobre o Cérebro), coordenado pelo neurologista
da FCM Fernando Cendes, que visa promover pesquisas em neurociências
e formar uma rede de cooperação entre instituições do Estado
de São Paulo. Ambas as iniciativas contaram com financiamento
da Fundação de Amparo à Pesquisa do Estado de São Paulo (Fapesp).
É sob este contexto que trabalha
Miranda, como “segmentador das imagens”, fazendo suas explorações
para desvendar achados cerebrais que possam levar à localização
de doenças. Em visão computacional, segmentação refere-se
ao processo de dividir uma imagem digital em múltiplas regiões
ou objetos, com a finalidade de simplificar a sua representação
e facilitar a sua análise. Com esta imagem é possível fazer
a definição espacial precisa de uma dada estrutura e, como
Miranda trabalha com imagens 3D, sua intervenção consiste
mais em analisar uma sequência de fatias integralmente.
Ele não está sozinho nos estudos
das imagens. No IC, Miranda divide esta tarefa com vários
pós-graduandos que trabalham para a resolução de outros tipos
de problemas, como realizar análise de assimetrias e de alterações
na anatomia do cérebro. O seu trabalho, comenta, é visto como
pré-requisito para outros. Mas o grupo do LIV já tinha experiências
anteriores com os softwares IBS e IVS, que segmentavam o cérebro
interativamente, e foi a partir deles que foi concebido o
Bia. Esta automação tem sido alcançada mediante um modelo
dos objetos, construído por um aprendizado supervisionado
pelo médico, o qual depende de uma base de treinamento com
imagens pré-segmentadas. Esse modelo é chamado Clouds, ou
modelo de nuvens, por ter esta aparência.
 Normalmente,
as ressonâncias atuais trabalham com imagens ponderadas de
T1. Com o Bia, a este processamento também se somarão as imagens
em T2 e em PD (proton density), nomenclaturas que exemplificam
modalidades de aquisição de imagens. A técnica da ressonância
magnética é usada principalmente para visibilizar a estrutura
cerebral. Normalmente,
as ressonâncias atuais trabalham com imagens ponderadas de
T1. Com o Bia, a este processamento também se somarão as imagens
em T2 e em PD (proton density), nomenclaturas que exemplificam
modalidades de aquisição de imagens. A técnica da ressonância
magnética é usada principalmente para visibilizar a estrutura
cerebral.
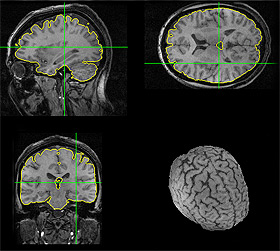
Todas as imagens do software
desenvolvido por Miranda, explica ele, são observadas como
se fossem volumes do cérebro humano. A cabeça está contida
nestes volumes, cabendo ao software ler esta imagem e apresentá-la
na tela em três cortes: o axial, o sagital e o coronal, que
denotam diferentes posições anatômicas, plotadas em três janelas
do computador. Uma quarta janela, à medida que é dissecado
virtualmente o cérebro, permite a visibilização da sua superfície.
A alimentação computacional
da estrutura do cérebro, que inclui o hemisfério esquerdo,
o direito e o cerebelo, futuramente será adaptada para as
estruturas subcorticais, estendendo-se ao núcleo caudado (localizado
nos núcleos da base, com um importante papel no controle motor)
e ao hipocampo (estrutura localizada nos lobos temporais,
considerada a principal sede da memória). “Nesta pesquisa
pretendemos que o processamento ocorra de maneira automática.
Apesar disso, reconhecemos que nem sempre podemos contar com
tal recurso, já que o processo pode estar sujeito a falhas.
Neste caso, a interpretação do médico será indispensável para
fazer as correções interativas.”
Miranda ainda descreve que
nesta atual fase de desenvolvimento do software, a de simulações,
um conjunto de imagens foram adquiridas pelo scanner da FCM,
que roda as abordagens da ferramenta em desktops convencionais
(computador de mesa). Mas fato é que elas também poderiam
ser utilizadas através de cluster (conjunto de computadores
que executam um determinado número de tarefas), a fim de obter
resultados mais rapidamente. O resultado automático do Bia
é comparado com o resultado do gabarito, decorrente da segmentação
interativa do especialista, de modo a avaliar a acurácia do
processo automático, verificando o quanto se aproxima do resultado
do especialista.
Neste caso, o Clouds define
para cada objeto uma imagem fuzzy, isto é, uma imagem de pertinência.
A diferença deste em relação a outros métodos empregados na
literatura, como o atlas (mapa probabilístico), é que a nova
abordagem evita o uso de registro não-rígido. Segundo Miranda,
o problema do registro é que ele tem de definir uma imagem
de referência, gerando um viés em relação à anatomia particular.
“Nosso método não precisa disso. A gente simplesmente separa
as imagens por grupos de alta similaridade e o ponto forte
do método é que ele explora a sinergia entre um modelo e um
algoritmo de delineamento, obtendo um resultado mais preciso.”
Este método do IC é inédito no mundo e a pesquisa sobre ele
trouxe várias contribuições na área, com divulgação em vários
periódicos internacionais, como no Journal of Matematical
Image Vision (JMIV), publicado em outubro deste ano.
Dissecação
Com a adoção desse modelo matemático do cérebro, o geométrico,
é possível fazer uma espécie de dissecação virtual. “Ao estudar
o cérebro, é preciso desmembrá-lo’, esclarece Falcão. Ele
deve, porém, atuar em conjunto com outro método, já em desenvolvimento
no IC, que separa os tecidos. As substâncias cinza e branca
do cérebro fazem parte dessas estruturas dissecadas.
Uma vez efetuada a dissecação,
a próxima parte inclui uma análise das assimetrias e das medidas
extraídas destes tecidos para criar uma base, que poderá ser
utilizada depois para fins de novas descobertas. Na vigência
de uma enfermidade, na tela do computador é apresentada tanto
a imagem de indivíduos normais quanto de indivíduos com alguma
patologia cerebral. A ideia é que o sistema consulte a base
de dados e descubra quais medidas conseguem discriminar melhor
as imagens daquela doença em relação às imagens normais, o
que pode ser usado para extrapolação.
A quantidade de dados, tanto
de imagens quanto de dados biológicos que poderão ser explorados
para esta finalidade, ninguém sabe “quem são” e “quais são”.
“Num primeiro momento armazenamos o máximo de informações.
As medidas, quando calculadas e transpostas para o sistema,
apontarão a probabilidade que aquele indivíduo tem para apresentar
determinada doença”. E o sistema automático pode dar este
indicativo, sugere Falcão. “Isso poderia auxiliar o diagnóstico
e dar suporte ao desenvolvimento de outras pesquisas em Neurologia”,
salienta.
No futuro poderão ser incorporados
a essa base de dados também a ressonância funcional, outros
tipos de modalidade de ressonância e até outras modalidades
de baixa resolução como as da medicina nuclear. A proposta
é registrar estas informações, que hoje estão disponíveis,
e os dados biológicos e de imagem. Isso, num segundo momento,
poderá ser usado para efetuar as inferências.
Processo
Todo o sistema é analisado através da tela do computador.
Como as imagens já são obtidas na forma digital, já que a
ressonância é digital, elas são armazenadas na base de dados
do projeto Cinapce. O papel do Bia, reforça Miranda, é ler
as imagens, processá-las e criar novas formas de enxergá-las.
Em outro trabalho, por exemplo, desenvolvido no ano passado,
a visibilização identificou regiões onde havia displasia (anomalia
no desenvolvimento de um órgão) cortical no cérebro, para
tentar confirmar o diagnóstico antes da cirurgia. Sem ela,
seria improvável identificar onde estaria a displasia, porque
a forma com que a imagem é adquirida na ressonância não permite
enxergá-la com nitidez.
As doenças que poderiam ser contempladas pelo uso desta ferramenta
seriam as epilepsias, que são o foco do Cinapce, e outras
doenças como o Alzheimer, a esquizofrenia e o Parkinson. “Contudo
ainda não sabemos definitivamente quais as patologias que
serão detectadas. Esta será uma etapa posterior. Mesmo assim,
o que queremos é ir um pouco além, não somente medir o volume
cerebral, mas ainda a diferença de forma e textura”, diz Falcão.
O grupo de Neuroimagem da Unicamp tem buscado conhecer a origem
dessas doenças, quais são as características que levam à ela
e saber a sua evolução.
Falcão revela que, se um paciente
com epilepsia não responde a um medicamento, então é removida
cirurgicamente a região do cérebro que está provocando este
distúrbio. Às vezes o procedimento é bem-sucedido e o paciente
é curado. Outras vezes não. Por quê? Há como saber anteriormente
se a cirurgia será eficaz? Não se sabe ao certo se o software
vai prever isso, todavia com certeza fornecerá um fundamental
suporte a esta pesquisa. “Particularmente, acredito que uma
vez sendo utilizado pelo grupo da Neurologia, a tendência
é que ele responda a estas perguntas. Infelizmente, com o
procedimento cirúrgico, alguns pacientes passam a ter certas
assimetrias, com ganho e perda de massa cinza (presente nos
neurônios) ou branca (axônios) em determinadas regiões, fatos
que podem estar associados à volta da crise.”
De acordo com Falcão, a confirmação
de uma doença pode ser até invasiva porém, quando não se sabe
localizar onde está o problema, como realizar a biópsia? O
primeiro passo então é a localização e o segundo a biópsia.
A pergunta hoje é: uma análise do cérebro através do computador,
mesmo não resolvendo o problema, pode indicar probabilidades
da existência de alguma doença? Muitos pacientes que se submetem
a um exame de rotina não são investigados de forma a detectar
o que está acontecendo dentro do seu cérebro. “Como os especialistas
não dispõem de softwares tridimensionais, o que os impede
de fazer uso de imagens processadas, simplesmente fazem a
aquisição das imagens em alguns cortes e os analisam visualmente,
imprimindo em filmes para poder respaldar o diagnóstico.”
No caso de displasia, se o
médico faz apenas cortes paralelos – os cortes tradicionais
da máquina –, ele pode não enxergá-la. O novo método proporciona
fazer o corte curvilinear, que acompanha a curvatura do cérebro.
Ali são mostradas informações relevantes que podem indicar
uma possível displasia. Esses novos processamentos aumentam
a sensibilidade diagnóstica, podem alertar para que o atendimento
seja mais cuidadoso e também ajudam o pesquisador a entender
melhor o cérebro.
|
|