O diferencial é a estabilidade da ligação da molécula com a proteína-alvo nos tumores, um avanço na busca de novas terapias oncológicas
Cientistas do Brasil e da Alemanha deram um passo importante na busca de um composto que pode gerar um novo tratamento contra o câncer. A molécula criada pelos cientistas é um potente inibidor da proteína MPS1, envolvida no crescimento de tumores sólidos. Os cientistas irão aprimorar a molécula e continuar com testes para saber como ela se comporta na inibição do crescimento de tumores sólidos em animais de laboratório e em células tumorais humanas. O estudo foi publicado hoje (16/2) no Journal of Medicinal Chemistry.
Os pesquisadores elegeram como alvo a MPS1 (Monopolar Spindle Kinase 1), uma proteína quinase relacionada a vários tipos de cânceres sólidos. As quinases são proteínas que regulam cascatas de reações químicas na célula, e, por isso, são consideradas alvos importantes para o desenvolvimento de medicamentos. A MPS1 é uma quinase que desempenha um papel crítico no controle da divisão celular. A superexpressão desta proteína tem sido associada a uma variedade de tumores sólidos, como os de mama, pâncreas, glioblastoma e neuroblastoma. O desenvolvimento de inibidores da MPS1 tem sido perseguido por cientistas visando o desenvolvimento de novas terapias oncológicas.
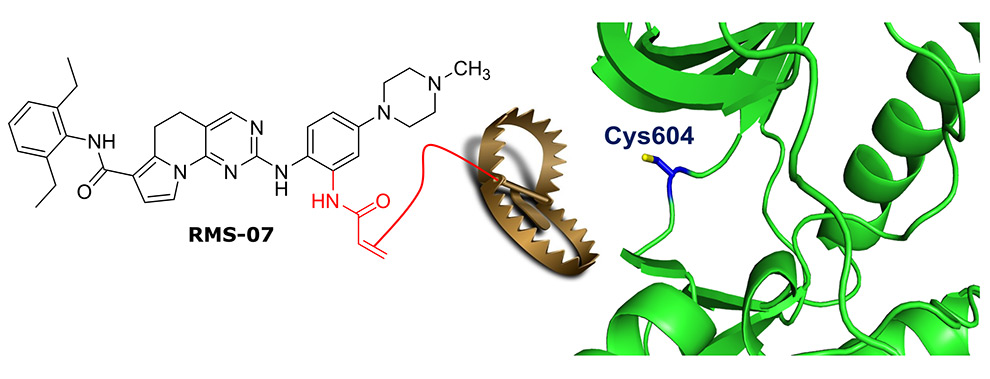
Nessa pesquisa, os cientistas testaram uma pequena molécula que se liga covalentemente à proteína da MPS1. “De maneira geral, os compostos se ligam a proteínas alvo de forma reversível, em equilíbrio dinâmico. A modificação que fizemos no composto faz com que ele se ligue de maneira definitiva. Isso traz vantagem para o desenvolvimento de um novo medicamento, pois há um prolongamento da ação inibitória na proteína”, explica Rafael Couñago, do Centro de Química Medicinal da Unicamp (CQMED), um dos autores do estudo.
Um aspecto importante no desenvolvimento de novos medicamentos é a forma como o composto se liga a proteína alvo. Essa característica influencia muito na concentração e eficácia do medicamento. A ligação covalente - quando há compartilhamento de elétrons entre os elementos -, se bem planejada, é uma característica desejada, pois é um tipo de ligação estável, garantindo que não haja deslocamento da molécula em relação a seu alvo. Isso poderia levar, por exemplo, à redução na frequência de administração do medicamento pelo paciente e mitigar o surgimento de mutações na proteína-alvo. Outros medicamentos que estabelecem este tipo de ligação são penicilina, omeprazol, aspirina, por exemplo.
Atualmente são conhecidos apenas oito medicamentos inibidores de quinases que se ligam de forma covalente. Nessa pesquisa, explorou-se uma característica pouco comum da MPS1: a presença de um resíduo do aminoácido cisteína em uma posição específica. Das mais de quinhentas proteínas quinases codificadas pelo genoma humano, apenas cinco têm tal característica. “Isto é favorável em termos de seletividade e potência quando se desenvolve uma molécula inibidora com potencial terapêutico, pois reduz-se em muito a chance de a nossa molécula se ligar a outras quinases”, explica Ricardo Serafim, um dos autores do estudo. Antigo pos-doutorando do CQMED, Serafim atualmente é pesquisador da Universidade de Tübingen, na Alemanha, onde realizou estágio com apoio da FAPESP
Para chegar à molécula inibidora covalente, os pesquisadores fizeram uma análise da estrutura tridimensional da MPS1, visando encontrar sítios promissores para o encaixe de moléculas. Em seguida, iniciaram a busca por moléculas inibidoras no Banco de Dados de Estrutura de Proteínas (PDB). Essa seleção inicial, juntamente com simulações computacionais, levou a um conjunto de três séries químicas promissoras.
Após dessa etapa, Serafim sintetizou aproximadamente trinta moléculas para testá-las em ensaios enzimáticos e identificou uma classe de compostos com o mecanismo de inibição desejada para a MPS1. A ligação covalente foi depois confirmada por meio de espectrometria de massa e cristalografia de raio-X. “Testamos a molécula em ensaios celulares e vimos que a molécula tem eficácia para tratar células cancerígenas”, explica Serafim.
Os experimentos que diferenciam inibidores covalentes de inibidores não covalentes podem ser bem complexos, requerendo diferentes expertises. “Trabalhamos bastante para poder comprovar que havia uma ligação covalente entre composto e alvo. Estar em um centro multidisciplinar capaz de desenvolver o ensaio enzimático, a espectrometria de massa e a cristalografia de raio-X ajudou muito a acelerar este processo”, ressalta Couñago. O CQMED é uma unidade da Empresa Brasileira de Inovação Industrial (EMBRAPII) e um Instituto Nacional de Ciência e Tecnologia (INCT) apoiado pela Fapesp, CAPES e CNPq sediado no Centro de Biologia Molecular e Engenharia Genética (CBMEG) da Unicamp.
Agora que chegaram na molécula inibidora de MPS1, os autores pretendem melhorar algumas de suas propriedades antes de iniciar os ensaios clínicos. “Tivemos sucesso na prova de conceito. Agora são necessários ajustes, principalmente em termos de reatividade, para que ela tenha ainda mais eficiência na inibição da proteína-alvo”, finaliza Serafim.